No processo de realização de estudos genéticos, muitas vezes encontramos amostras de RNA insuficientes, por exemplo, para estudar minúsculos tumores orais anatômicos, mesmo amostras de células únicas e amostras de mutações genéticas específicas que são transcritas em níveis muito baixos em células humanas.Claro, para o teste COVID-19, se os swabs não estiverem no lugar certo ou não forem suficientes durante a amostragem, o tamanho da amostra será muito baixo, por isso a Comissão de Saúde e Planejamento Familiar saiu há dois dias e passou no teste e, se o amostrador de ácido nucleico não colheu seis amostras, você pode denunciá-lo.
A sensibilidade do reagente é importante porque temos este ou aquele problema, então o que podemos fazer para melhorar a sensibilidade do RT-PCR?
Antes de discutirmos possíveis soluções, vamos mencionar duas grandes complicações com a situação que acabamos de mencionar.
Em primeiro lugar, nos preocupamos com a perda de RNA quando temos apenas algumas populações de células em nossa amostra.Se métodos tradicionais de separação e limpeza forem usados, como método de coluna ou método de precipitação de ácido nucleico, há uma grande possibilidade de que algumas amostras sejam perdidas.Uma solução é adicionar uma molécula transportadora, como tRNA, mas mesmo assim, não há garantia de que nosso experimento de recuperação esteja OK.
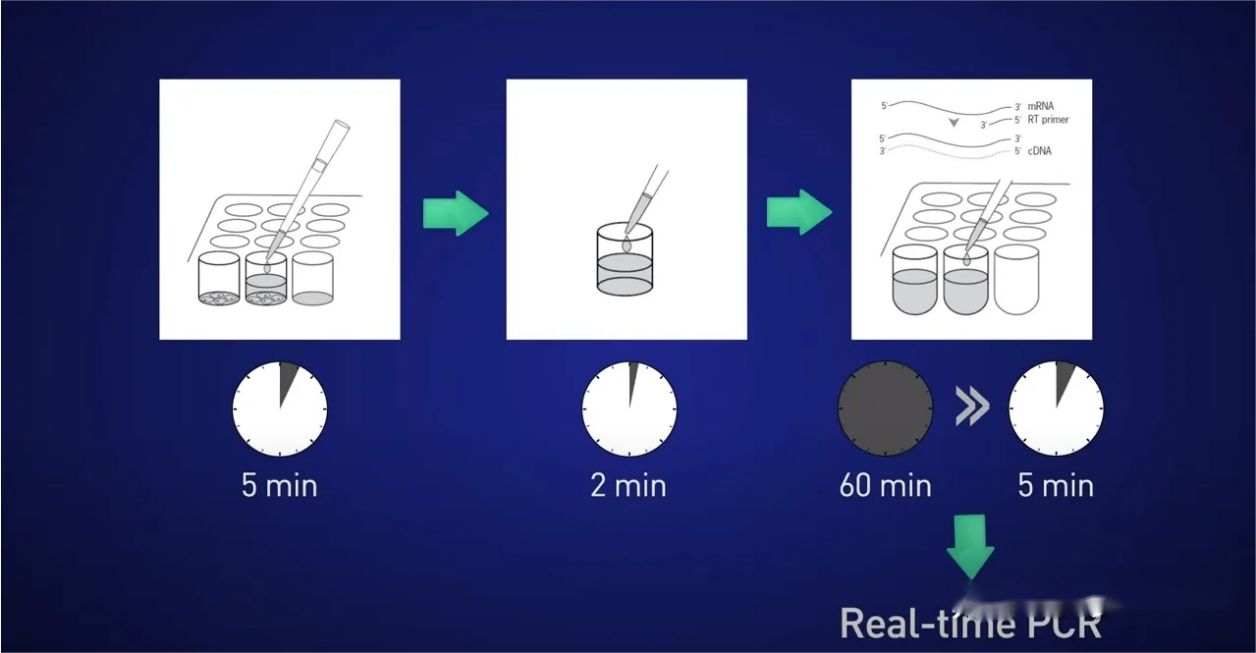
Então, qual é a melhor maneira?Uma boa opção para células cultivadas ou amostras microanatômicas é usar a lise direta.
A ideia é dividir as células por 5 minutos, liberar o RNA na solução, depois parar a reação por 2 minutos, depois adicionar o lisado diretamente na reação de transcrição reversa para que nenhum RNA seja perdido e, por fim, colocar o cDNA resultante diretamente na reação em tempo real.
Mas e se, devido a um ponto de partida limitado ou a uma pequena quantidade de expressão do gene alvo, pudermos reciclar todo o RNA e ainda não fornecer modelos suficientes para obter um bom sinal em tempo real?
Nesse caso, a etapa de pré-amplificação pode ser muito útil.
O seguinte é um esquema para aumentar a sensibilidade após a transcrição reversa.Antes de começar, precisamos perguntar a jusante quais alvos nos interessam, de modo a projetar primers específicos para esses alvos para pré-amplificação.
Isso pode ser obtido criando um primer misto com até 100 pares de primers e um ciclo de reação de 10 a 14 vezes.Portanto, um Master Mix projetado especificamente para este requisito é necessário para pré-amplificar o cDNA obtido.
A razão para definir o número de ciclos entre 10 e 14 é que esse número limitado de ciclos garante a aleatoriedade entre os vários alvos, o que é crucial para pesquisadores que precisam de informações moleculares quantitativas.
Após a pré-amplificação, podemos obter uma grande quantidade de cDNA, de modo que a sensibilidade de detecção no back-end seja bastante aprimorada, e podemos até diluir a amostra e realizar várias reações de PCR em tempo real para eliminar possíveis erros aleatórios.
Horário de postagem: 11 de abril de 2023